使用基因分型定序鑑定 Cinnamomum kanehirae Hayata 和 C. Camphora (L.) Presl 之間的雜交和基因滲入
圖
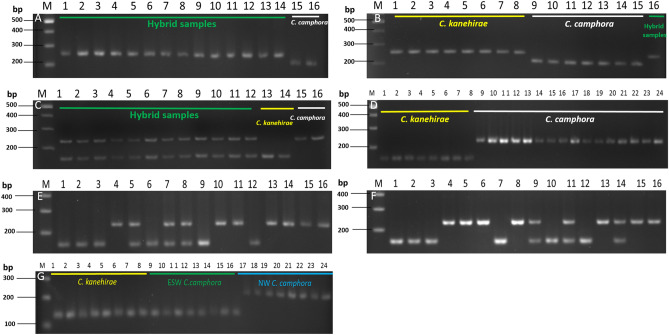
圖 1
InDel,…的凝膠電泳
圖1
InDel、ZS_CK InDel_1(粒線體DNA)( A,B )、JLH2_CK…的凝膠電泳
圖1 InDel、ZS_CK InDel_1(粒線體DNA)(A,B)、JLH2_CK InDel_4(共顯性)(C–F)凝膠電泳)和 ZS_CK InDel_2(葉綠體 DNA)(G)。 M:100 bp DNA 梯子。 (A) 泳道 1-14:雜種,泳道 15-16:香樟。 (B) 泳道 1-8:C. kanehirae,泳道 9-15:C. Camphora,泳道 16:雜交種。 (C) 泳道 1-12:雜種,泳道 13 和 14:C. kanehirae,泳道 15 和 16:C. Camphora。 (D) 泳道 1-8:C. kanehirae,泳道 10-24:C. Camphora。 (E) 泳道 1:C. kanehirae,泳道 2-14:雜種,泳道 15 和 16:C. Camphora。 (F) 泳道 1:C. kanehirae,泳道 2-14:雜種,泳道 15 和 16:C. Camphora。 (G) 泳道 1-8:C. kanehirae,泳道 9-16:ESW C. Camphora。泳道 17-24:西北樟腦。所使用的樣本列於線上補充表 S5 中。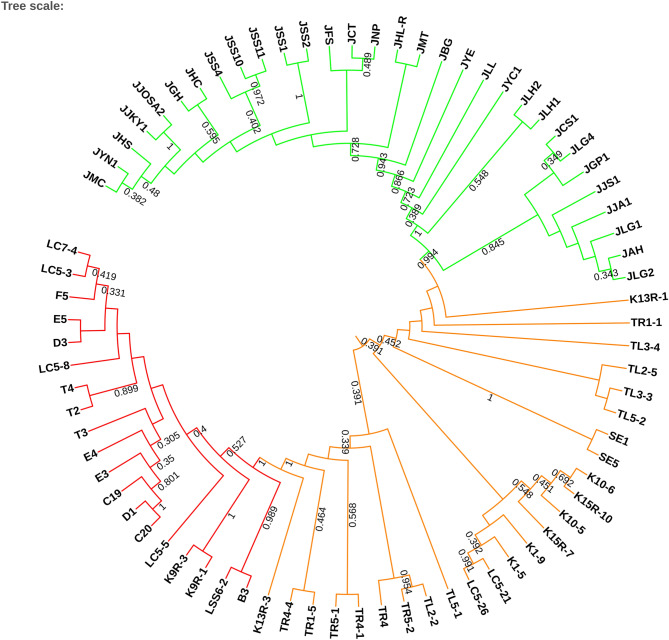
圖2
鄰接樹基於808 …
圖2
基於808 SNP的鄰接樹。顯示了 1000 個重複的 Bootstrap 值...
圖 2 基於 808 個 SNP 的鄰接樹。 1000 個重複的引導值顯示在節點上。樹枝上的顏色是按簇著色的。 C. kanehirae 為紅色,假定的雜交種為橘色,C. Camphora 為綠色。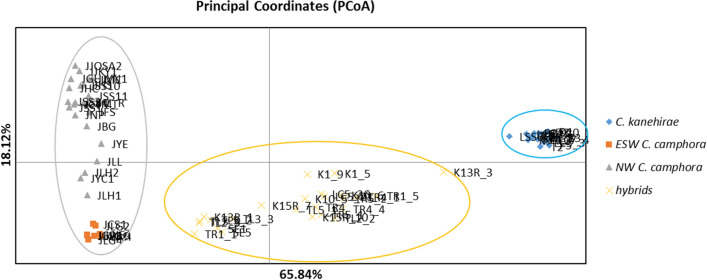
圖3
主座標分析(基於PCoA)…
圖3
基於C的Nei成對遺傳距離的主座標分析(PCoA)…
圖3 基於主座標分析(PCoA) C . kanehirae、C. Camphora 及其雜種(75 個樣本)的Nei 成對遺傳距離。每個橢圓內的彩色符號如圖例所示。範例如線上補充表 S4 所示。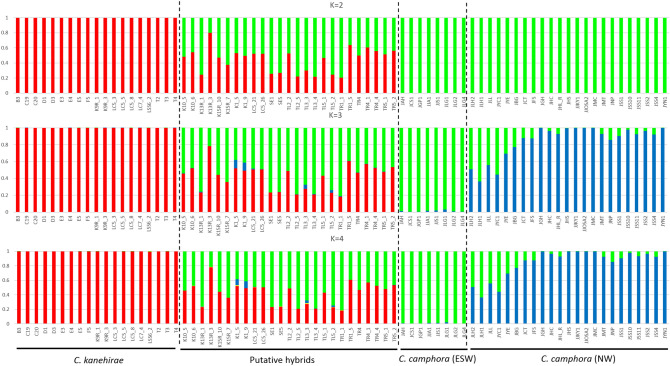
圖4
辨識與分類雜交種。結構結果顯示基因組的比例...
圖 4 雜交種的鑑定與分類。 STRUCTURE 結果顯示源自每個推斷簇的每個個體的基因組比例 (K = 2);紅色是簇 1 (C. kanehirae),綠色是簇 2 (C. Camphora);雜交種混合了紅色和綠色。 K = 3的結構,將綠色簇的23份材料分為藍色簇。所有 ESW 樟樹在綠色簇中都具有高 Q 值。 K = 4的結構,在C. kanehirae和假定的雜種中,只有紅色簇被分為黃色簇。虛線根據形態特徵和系統發育分析進行分類。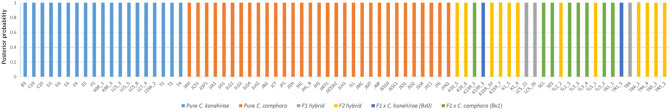
圖 5
NewHybrids 結果假定. ..
圖5
NewHybrids 結果以及所有75 個人的假定譜系和後驗機率...
圖5 NewHybrids 結果以及所有75 個人的假定譜係以及後驗機率不同色條中給定類別的機率。